วันนี้ (7 ก.พ.2566) นพ.ศุภกิจ ศิริลักษณ์ อธิบดีกรมวิทยาศาสตร์การแพทย์ กล่าวถึง สถานการณ์การเฝ้าระวังสายพันธุ์โควิด-19 และสายพันธุ์ที่เฝ้าติดตามในประเทศไทย ว่า กรมวิทย์ฯ ร่วมกับเครือข่ายห้องปฏิบัติการ ติดตามการเปลี่ยนแปลงสายพันธุ์เชื้อไวรัส SARS-CoV-2 พบเชื้อไวรัสมีการกลายพันธุ์อย่างต่อเนื่อง
ปัจจุบันองค์การอนามัยโลกกำลังให้ความสำคัญกับการติดตามโอมิครอน 4 สายพันธุ์ จากพื้นฐานของข้อมูลการเพิ่มความชุกหรือความได้เปรียบด้านอัตราการเติบโตเมื่อเทียบกับสายพันธุ์อื่น ๆ และการกลายพันธุ์ในตำแหน่งที่เกี่ยวข้องกับการได้เปรียบในการก่อโรค โดยช่วงเดือน ม.ค.2566 ที่ผ่านมา พบข้อมูลสายพันธุ์ในฐานข้อมูลสากล GISAID ดังนี้
- BF.7 จำนวน 1,147 ตัวอย่าง ร้อยละ 4.6
- BQ.1 และลูกหลาน จำนวน 11,674 ตัวอย่าง ร้อยละ 46.9 รวมถึง BQ.1.1 7,189 ตัวอย่าง ร้อยละ 28.9
- BA.2.75 และลูกหลาน จำนวน 3,473 ตัวอย่าง ร้อยละ 13.9 รวมถึง BA.2.75.2 35 ตัวอย่าง ร้อยละ 1 และ CH.1.1 1,672 ตัวอย่าง ร้อยละ 6.7
- XBB และลูกหลาน จำนวน 4,049 ตัวอย่าง ร้อยละ 16.3 รวมถึง XBB.1.5 3,005 ตัวอย่าง ร้อยละ 12.1
"โอมิครอน" สายพันธุ์หลักที่แพร่กระจายอยู่ในไทย
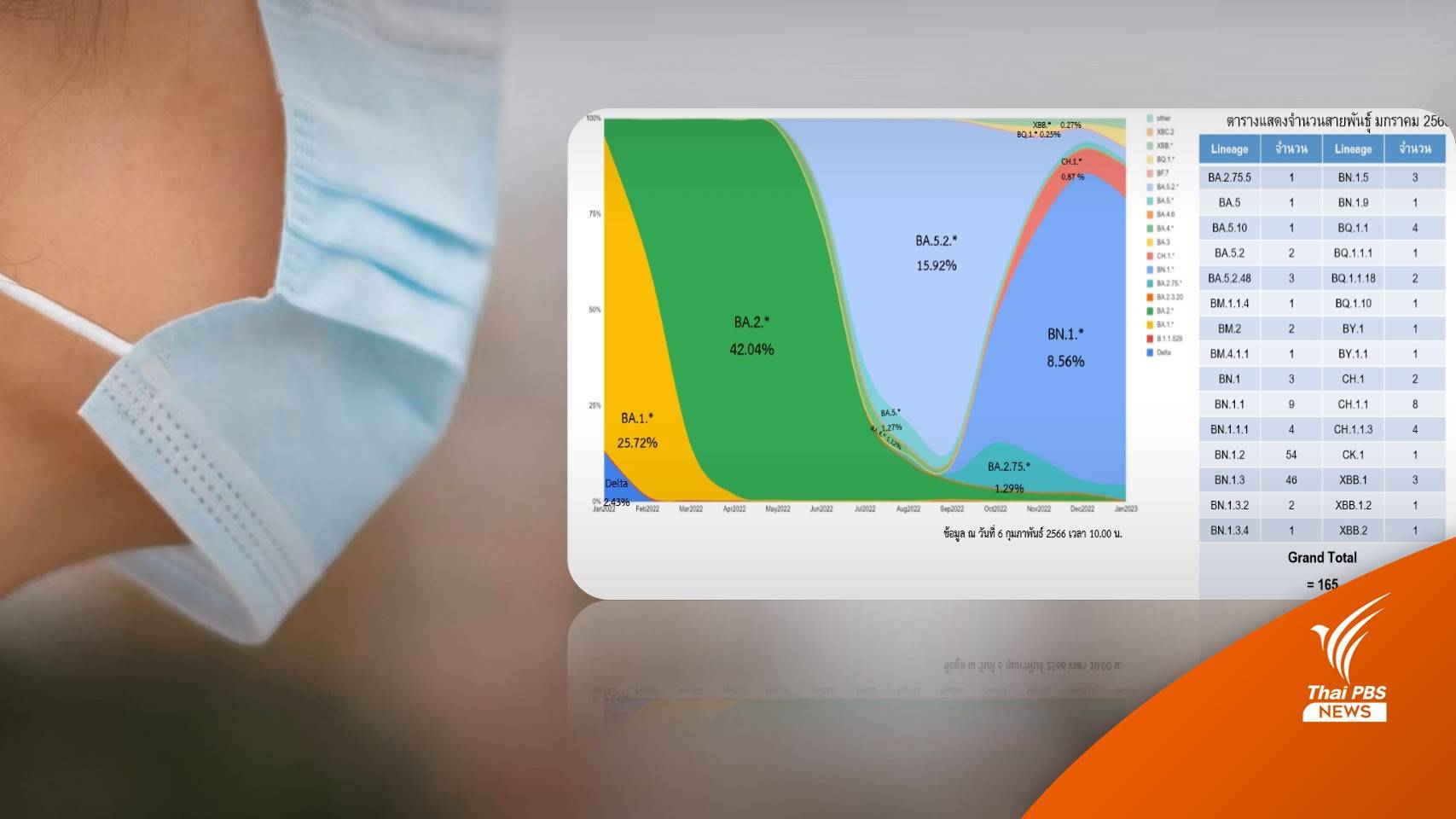
นพ.ศุภกิจ กล่าวว่า สถานการณ์สายพันธุ์โควิด-19 ในประเทศไทย ตั้งแต่ต้นปี 2565 พบสายพันธุ์เดลตา ถูกแทนที่ด้วยสายพันธุ์โอมิครอน สายพันธุ์ย่อยต่าง ๆ ได้แก่ BA.1, BA.2, BA.4, BA.5 และสายพันธุ์ย่อยอื่น ๆ ในตระกูล ซึ่งปัจจุบันสายพันธุ์โอมิครอนเป็นสายพันธุ์หลักที่แพร่กระจายอยู่ในประเทศไทย
ผลการเฝ้าระวังสายพันธุ์เชื้อก่อโรคโควิด-19 ตั้งแต่ต้นปี 2566 จากผลการตรวจแบบ SNP/Deletion จำนวน 689 คน พบสัดส่วนสายพันธุ์หลัก คือ สายพันธุ์ BA.2.75.* คิดเป็นร้อยละ 88.5 โดยผลการตรวจแบบ SNP/Deletion จำนวน 94 คน

ในรอบสัปดาห์ที่ผ่านมาระหว่างวันที่ 28 ม.ค. - 3 ก.พ.2566 พบสัดส่วนสายพันธุ์ BA.2.75.* เป็นหลักในกลุ่มผู้ติดเชื้อ คิดเป็นร้อยละ 87.2 จำนวน 82 คน และพบสายพันธุ์ BA.4/BA.5 คิดเป็นร้อยละ 8.5 จำนวน 8 คน
โดยสัดส่วนสายพันธุ์ BA.2.75.* ร้อยละ 85.1 พบในกลุ่ม ผู้ติดเชื้อในประเทศ ซึ่งเพิ่มขึ้นอย่างต่อเนื่องตั้งแต่เริ่มสถานการณ์ระบาดในเดือน ก.ย.2565 และกลายเป็นสายพันธุ์หลักที่ระบาดในประเทศแทนที่สายพันธุ์ BA.5
สำหรับผลการถอดรหัสพันธุกรรมแบบทั้งตัว (Whole genome sequencing) ของตัวอย่างในประเทศไทยจนถึงปัจจุบัน พบสายพันธุ์ BA.2.75 และลูกหลานของ BA.2.75 (BA.2.75.*) เช่น BA.2.75.2, BA.2.75.5, BA.2.75.5.1 (BN.1), BA.2.75.5.1.3 (BN.1.3), BA.2.75.3.4.1.1.1.1 (CH.1.1) จำนวนเพิ่มขึ้นอย่างต่อเนื่องตั้งแต่เริ่มพบในประเทศไทยเมื่อปลายเดือน มิ.ย.2565
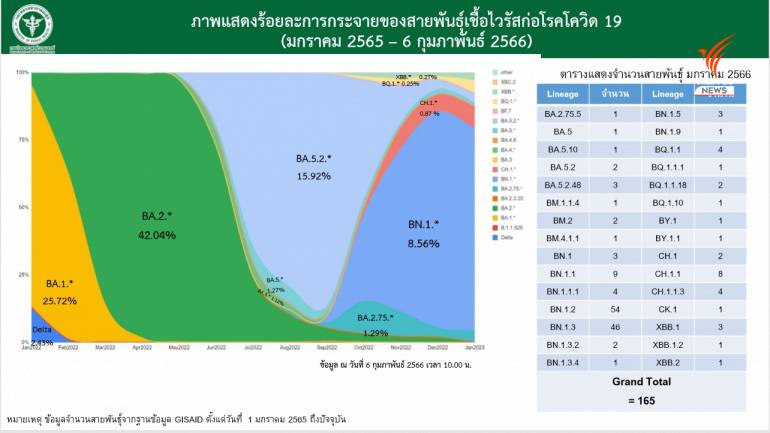
จากการวิเคราะห์ข้อมูลสายพันธุ์ที่เผยแพร่ในรอบ 4 เดือน (ต.ค.2565 - ม.ค.2566) พบอุบัติการณ์สายพันธุ์ BA.2.75.* ร้อยละ 73.8 ซึ่งรวมถึง BN.1.* ร้อยละ 59 และ CH.1.1.* ร้อยละ 6.8 โดยสายพันธุ์ที่พบมีสัดส่วนสูง ได้แก่ BN.1.* ซึ่งมีสัดส่วน ของสายพันธุ์ BN.1.3.* สูงที่สุด คิดเป็นร้อยละ 82.4
ในขณะที่สายพันธุ์ BA.5.2 ซึ่งเดิมเคยเป็นสายพันธุ์หลักในประเทศไทยมีสัดส่วนลดลง คิดเป็นร้อยละ 17.9 และในเดือน ม.ค.2566 ข้อมูลการกระจายสายพันธุ์หลักที่พบในประเทศไทย ยังคงเป็นสายพันธุ์ BN.1* ร้อยละ 74.5
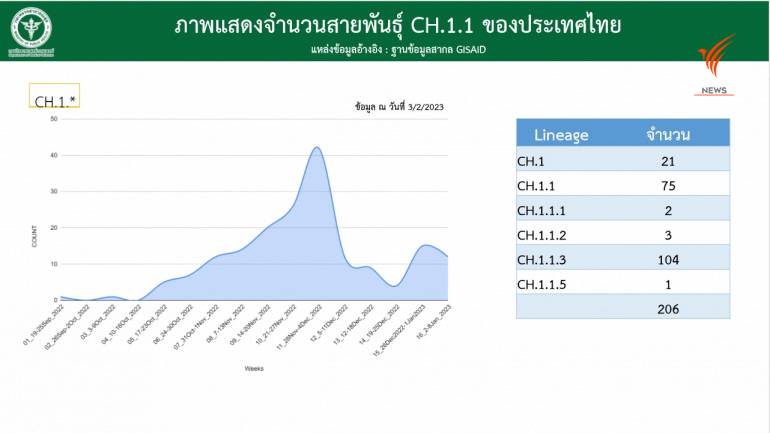
CH.1.1 และสายพันธุ์ย่อย มีแนวโน้มหลบภูมิได้ดี
สำหรับสายพันธุ์ CH.1.1 เป็นสายพันธุ์ย่อยของ BA.2.75 (BA.2.75 + R346T, K444T, L452R, และ F486S) สามารถหลบภูมิคุ้มกันได้พอสมควร พบรายงานครั้งแรกในอินเดีย เมื่อวันที่ 8 ก.ค.2565 และแพร่กระจายไปทั่วโลกตั้งแต่เดือน พ.ย.2565 ในเดือน ม.ค.2566 ที่ผ่านมา พบสายพันธุ์ CH.1.1 และสายพันธุ์ย่อย กว่าร้อยละ 6 ของข้อมูลจากทั่วโลก (ข้อมูล ณ วันที่ 30 ม.ค.2566)
และพบมากที่สุดในสหราชอาณาจักร เดนมาร์ก และสิงคโปร์ โดยสายพันธุ์ CH.1.1 มีการกลายพันธุ์บริเวณส่วนหนามที่สำคัญ คือ K444T, L452R, N460K, และ F486V ซึ่งทำให้หลบภูมิคุ้มกันจากการติดเชื้อตามธรรมชาติ หรือ จากการฉีดวัคซีนได้ดี มีคุณสมบัติดื้อต่อแอนติบอดีสังเคราะห์ Evusheld และ Bebtelovimab
สำหรับประเทศไทย พบรายงานครั้งแรก เมื่อ พ.ย.2565 สายพันธุ์ CH.1.1 และสายพันธุ์ย่อย คิดเป็นกว่าร้อยละ 7.3 ของข้อมูล sequence ที่เผยแพร่ในเดือน ม.ค.2566 ที่ผ่านมา
ส่วนสายพันธุ์ XBB.1.5 ที่ระบาดในอเมริกา ยังไม่พบในไทย
นพ.ศุภกิจ กล่าวว่า ขอให้ความมั่นใจว่ากรมวิทยาศาสตร์การแพทย์ และเครือข่าย ยังคงเฝ้าระวังติดตามการกลายพันธุ์ของเชื้อ SARS-CoV-2 อย่างต่อเนื่อง และเผยแพร่บนฐานข้อมูลสากล GISAID อย่างสม่ำเสมอ ซึ่งมีการแชร์ข้อมูลจากนักวิทยาศาสตร์ทั่วโลก
ทั้งนี้ เพื่อติดตามผลกระทบจากสายพันธุ์ย่อยของสายพันธุ์น่ากังวล ความรุนแรงของโรค หรือคุณสมบัติของอื่น ๆ ที่เกี่ยวข้อง ของเชื้อไวรัส เป็นข้อมูลสนับสนุนการออกแบบการรักษา การให้ยาต้านไวรัส หรือแอนติบอดีสังเคราะห์